Datentypen, Datenformate und der ganze Kram...¶
In dieser Lektion wollen wir nochmal einen Schritt zurücktreten und betrachten, womit wir es zu tun haben, wenn wir von Daten sprechen. Dabei gibt es unterschiedliche Perspektiven:
Datentypen¶
In Python werden Daten durch bestimmte Datentypen repräsentiert. Es gibt primitive Datentypen, die es auch außerhalb der Pythonwelt bekannt sind (z.B. float, int usw.). Andere Datentypen gibt es nur in Python (z.B. dict).
Dateien¶
Außerhalb von Python existieren Daten typischerweise in Dateien. Was muss man über diese wissen?
- Kodierung: Um eine Datei lesen zu können, musst Du die "Kodierung" kennen. Uns vertraut ist uns z.B. die Kodierung als Text.
- Struktur: Um den Inhalt einer Datei interpretieren oder verarbeiten zu können, musst Du ihre Struktur kennen - wie sind die Daten organisiert? Zum Beispiel können die Daten in Spalten organisiert sein (Tabelle): Jede Spalte hat dann einen Datentyp, der sich aber nicht immer vollkommen intuitiv erschließt.
Format¶
Meist spricht man von einem Format als einer Kombination aus einer Kodierung und einer Struktur. Dieses Format wird manchmal, aber nicht immer, durch die Dateiendung angezeigt. Beispiele:
.csv: Die Endungcsvzeigt eine Tabelle an, die als Text kodiert ist. Wie die Spalten getrennt sind und was für Datentypen die Spalten enthalten, sagt uns die Endung nicht..xlsx: Diese Endung ("Excel-Datei") zeigt eine Tabelle an, die in einem binären Spezialformat kodiert ist.
Spröde?¶
Ja! Dieser einführende Textbatzen signalisiert bereits, dass wir es mit einer sehr spröden Materie zu tun haben. Aber wer mit Umweltdaten arbeitet, muss sich tagtäglich mit genau solchen Fragen beschäftigen...also los!
Datentypen¶
Aufgabe¶
Ihr habt in Python bereits viele Datentypen kennengelernt. Versucht (mit Euren Nachbar:innen?) eine möglichst vollständige Liste der Euch bekannten Datentypen in Python zusammenzustellen, mit einem Beispiel für jeden Datentyp. Tipp: Nutzt die type-Funktion. Und Ihr dürft auch Pakete importieren.
# Beispiel integer
type(1)
Lösung¶
import datetime as dt
data = [True, 1, 1., "umwelt", [92, 93, 94], {"a": 1, "b": 2}, dt.datetime.now(),dt.datetime.now,
dt.datetime.now()-dt.datetime.now(), range(5)]
for item in data:
print("- %s is %s" % (item, type(item)))
Datentypen umwandeln (type conversion)¶
Primitive Datentypen können wir teils ineinander umwandeln. Man nennt dies "type conversion".
# int zu float
float(5)
# float zu int
int(5.0)
# str zu int
int("5")
# str zu float
float("5.0")
# bool zu float
float(True)
# list zu str
str([1,2,3])
# float zu bool (alles außer 0 ist True)
bool(5.)
Warum interessiert uns das?¶
Die Entscheidung, welchen Datentyp eine Variable annimmt, trifft Python oft im Hintergrund, ohne dass wir uns darum kümmern müssen. Manchmal müssen wir uns aber bewusst machen, womit wir es zu tun haben...
Beispiel: Präzision¶
Der vorliegende Fall stellt unser gewohntes Verständis von True und False auf den Kopf...
(0.3 - 0.2 - 0.1 ) == 0
0.3 - 0.2 - 0.1
Der Grund für dieses überraschende Ergebnis: Computer speichern Zahlen nicht im Dezimalsystem, sondern im Binärsystem ab. Bestimmte Dezimalzahlen lassen sich so nicht präzise abbilden. Man sich vorstellen, dass so etwas in einem Programm zu schwer auffindbaren Fehlern führen kann...
Beispiel: Indizierung¶
Die Indizierung einer Liste oder eines Arrays funktioniert nur mit Integern.
nottodo = ["Frühzeitig an die Rente denken.",
"Sich an Kleinkunstprojekten beteiligen.",
"Aus Langeweile ein Detektivbüro gründen"]
nottodo[1.]
nottodo[1]
Beispiel: Gefährliche Zeiger¶
Eine Liste ist eigentlich eine übersichtliche Angelegenheit.
bewerberinnen = ["alf", "eric", "bart", "kyle", "stan", "kenny"]
Wir wollen nun aus den Bewerber:innen (?!) eine Vorauswahl treffen. Dazu erzeugen wir aus der Liste bewerberinnen eine Liste namens vorauswahl. alf und bart fliegen raus, sie passen nicht ins gewünschte Profil.
vorauswahl = bewerberinnen
vorauswahl.remove("alf")
vorauswahl.remove("bart")
vorauswahl
alf hat allerdings den Rechtsweg bestritten und sich eingeklagt. Wir sind also gezwungen, uns die ursprüngliche Liste der bewerberinnen nochmal anzuschauen..doch...auweia:
bewerberinnen
Wie ist das passiert?? Das geht doch sonst auch:
a = 1
b = a
b = 2
a, b
Aber eben nicht mit Listen oder Arrays. Diese werden vom System nämlich als "Zeiger" repräsentiert. Was das ist? Oje, das besprechen wir ein anderes Mal. Wie man das Problem vermeiden kann?
liste1 = [1,2,3]
liste2 = liste1.copy()
liste2.remove(2)
liste1
Beispiel: Datumsangaben¶
Nehmen wir an, Ihr habt eine Textdatei eingelesen und erhaltet folgende Zeichenkette zurück:
x = "01/02/03"
x
Tja...wahrscheinlich handelt es sich um ein Datumsformat. Das sollte sich hoffentlich aus dem Kontext oder noch besser der Dokumentation ergeben. Aber was ist jetzt Tag, Monat und Jahr? In den USA wäre es wohl der 2. Januar 2003, in Großbritannien eher der 1. Februar 2003 - manche Schlaumeier:innen schreiben vielleicht das Jahr nach vorne, weil sie das mal "irgendwo so gesehen haben". Klar ist: Die Interpretation der Zeichenkette als Datum ist kontextabhängig.
# the American way of style
date = dt.datetime.strptime(x, "%m/%d/%y")
str(date), date
Aufgabe: Datumsangabe I¶
Interpretiere den obenstehenden Datumsstring x auf britische Weise und formatiere das resultierende Datum y mit y.strftime("...") so, dass eine Datumsangabe im Stil von 24. Dezember 2021 entsteht. Hier kannst Du sehen, wie Du den Format-String schreiben musst, um das gewünschte Ergebnis zu erzielen.
Lösung¶
# the British way of style
y = dt.datetime.strptime(x, "%d/%m/%y")
y
y.strftime("%d. %B %Y")
Aufgabe: Datumsangaben II¶
Aber auch bei folgender Integer handelt es sich um eine Datumsangabe.
x = 1640304000
Hast Du eine Idee, wie hier das Datum kodiert ist und wie man es mit Hilfe des datetime-packages in eine verständliche Zeitangabe umwandeln kann?
Lösung¶
Es handelt sich um einen so genannten UNIX (POSIX) Timestamp (Zeitstempel): ein weit verbreitetes Format, in dem Datum und Zeit als die Zahl der Sekunden dargestellt wird, die seit dem 1. Januar 1970 vergangen sind.
dt.datetime.utcfromtimestamp(x)
Überprüfung der Richtigkeit:
dt.datetime.utcfromtimestamp(x) - dt.timedelta(seconds=x)
Daten brauchen Kontext!¶
Der letzte Teil über Datumsangaben hat uns bewusst gemacht, dass die Interpretation und Nutzung von Daten immer auch einen Kontext bzw. eine Dokumentation erfordert. Dieses Zusammenspiel zwischen Typ, Struktur und Format wollen wir in der Folge weiter vertiefen.
Text vs. Nicht-Text¶
Die am weitesten verbreitete Kodierung ist der Text. Textdateien kann man in einem Texteditor wie notepad++ betrachten und erkunden. Auf diese Weise erhält man im besten Fall einen Eindruck über die Struktur der Daten.
Schau Dir z.B. mal die Datei data/data.txt an. Aha, spannend: Zahlen von 0 bis 19, organisiert in Zeilen und Spalten. Man kann diese z.B. mit numpy.loadtxt einlesen und erhält einen zweidimensionalen Array:
import numpy as np
x = np.loadtxt("data/data.txt")
x
type(x)
Aufgabe: int statt float¶
Wie könnt Ihr sicherstellen, dass diese Datei als Array von int-Werten, nicht float-Werten eingelesen wird?
Lösung¶
x = np.loadtxt("data/data.txt", dtype="int")
x
Nicht-Text-Aufgabe¶
Öffnet nun einmal die Datei data/data.npy in Eurem Texteditor. Was seht Ihr? Bei mir (Ubuntu, gedit) Folgendes:
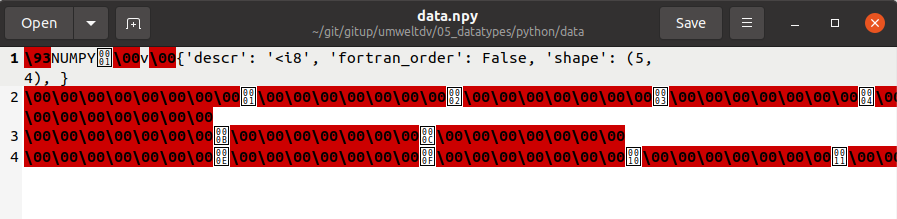
Das bezeichnet man landläufig wohl als "Kraut und Rüben". Aber ist gibt immerhin ein kleines Textfragment, das uns erklärt, womit wir es zu tun haben: einer binären Numpy-Datei. Erfahrene Nutzer:innen wissen, was sie zu tun haben. Andere müssen die Suchmaschine anschmeißen: Wie könnt Ihr diese Datei lesen?
Lösung¶
Ich bin mal wieder bei stackoverflow fündig geworden.
x = np.load("data/data.npy")
x
Fazit¶
Die gleichen Daten können als Text oder binär gespeichert werden. Was genau "binär" heißt, werden wir hier nicht behandeln. Sagen wir mal so: Wenn es in unserem Texteditor wie Kraut und Rüben aussieht, haben wir es wahrscheinlich mit einem Binärformat zu tun.
Um eine Binärdatei zu lesen, benötigt man eine Funktion/ein Package/eine Software, welche die Datei "versteht" (Kodierung und Struktur). Gelegentlich steht man als Wissenschaftler:in vor einer nicht dokumentierten Binärdatei und weiß nicht, wie man diese lesen soll - blöd!
Dabei kann durchaus sinnvoll sein, Daten in einem Binärformat abzuspeichern, z.B. um den Speicherbedarf zu reduzieren oder die Lesegeschwindigkeit zu erhöhen. In manchen Bereichen sind binäre Formate die Regel, nicht die Ausnahme (z.B. bei Bildern, Geodaten, uvm.).
Dateiendungen¶
Eine Dateiendung wie .csv, .txt, .png, oder .jpg zeigt uns zwar an, womit wir wir es zu tun haben, ist aber ansonsten unerheblich. Die Dateiendung wird erst dadurch relevant, dass sie auf unseren Rechnern teils mit bestimmten Anwendungen verknüpft ist.
Aufgabe: Endung manipulieren¶
Probier es mal anhand der Datei ../data/06_Matjpg.jpg aus (einer Bilddatei).
- Lege bitte zunächst eine Kopie an und nenne diese
../data/logo.jpg. - Ein Doppelklick auf die Datei sollte auf Deinem System nun das Unilogo in irgendeinem Bildbetrachter öffnen.
- Ändere die Dateinamen von
logo.jpgzulogo.txt - Versuche nun die Datei zu öffnen. Was passiert?
- Wie kannst Du sie trotzdem öffnen?
Lösung¶
Wenn auf Deinem System .txt-Dateien mit einem Texteditor verknüpft sind, wird sich logo.txt in Deinem Texteditor öffnen. Wenn Du aber die Datei im Bildbetrachter öffnest (Shift + rechte Maustaste > "Öffnen mit..."), wird dennoch das Bild angezeigt. Wir können das auch direkt in Python zeigen:
import matplotlib.image as mim
import matplotlib.pyplot as plt
im = mim.imread("../data/logo.txt")
plt.imshow(im)
_ = plt.axis("off")
Dateien mit bestimmter Endung suchen¶
Man kann zwar logo.jpg in logo.txt umbennen - sollte es aber lassen. Denn wie gesagt: Die Endung signalisiert uns, womit wir es zu tun haben. Auf diese Weise können wir z.B. ganze Verzeichnisse nach bestimmten Dateitypen durchforsten, um diese nacheinander zur verarbeiten. Dazu gibt es das geniale Modul glob.
import glob
Wir haben für Dich Ostereier im Verzeichnis data versteckt. Diese werden durch die Dateiendung .ei angezeigt und enthalten jeweils eine Farbe. Wir können jetzt mit glob alle Eier im Verzeichnis finden. Dafür nutzen wir sogenannte "Wildcards".
Zunächst suchen wir nur alle Eier im Verzeichnis data.
for name in glob.glob('data/*.ei'):
print(name)
Nun suchen wir auch in allen Unterverzeichnissen nach Eiern. Man nennt dies auch "rekursive" Suche.
for name in glob.iglob('data/**/*.ei', recursive = True):
print(name)
Aufgabe: Farbe bekennen¶
Jede .ei Datei enthält die Farbe des Eies (als String). Anstatt den Dateinamen zu drucken: Lies die Datei ein und drucke die Farbe. Wie liest man eine Textdatei?
f = open(pfadname)
txt = f.read()
f.close()Lösung¶
for name in glob.iglob('data/**/*.ei', recursive = True):
f = open(name)
txt = f.read()
f.close()
print(txt)
print("(Happy Christmas!)")
Typische Probleme mit Tabellen¶
Nur weil man Textdateien im Editor betrachten kann, ist das Einlesen der Daten in Python nicht automatisch "leicht". Aber wir können auf diese Weise ggf. Rückschlüsse auf die Struktur der Daten ziehen und so Probleme lösen.
Aufgabe: komischer Spaltentrenner, kein Header¶
Der DWD stellt in diesem Verzeichnis die Daten der Webcam an der Station Hohenpeißenberg zur Verfügung. Dazu gibt es auch die Metadaten der Webcam (also Infos über das Instrument).
Lies die Metadaten in der jüngsten Webcam-Aufzeichnung als DataFrame ein. Schönes Feature: Du brauchst die Datei gar nicht runterzuladen, sonden kannst gleich die URL als Dateipfad verwenden: https://opendata.dwd.de/weather/webcam/Hohenpeissenberg-S/Hohenpeissenberg-S_latest.txt
dateipfad = "https://opendata.dwd.de/weather/webcam/Hamburg-SW/Hamburg-SW_latest.txt"
import pandas as pd
Lösung¶
pd.read_table(dateipfad, sep="|", header=None)
Aufgabe: Typberatung für Spalten¶
Die Datei data/schnee_potsdam.csv enthält eine Tabelle mit Schneemessungen an der DWD-Station Potsdam. Die Spalte hoehe steht für die gemessene Schneehöhe (in cm), die Spalte weq für das Schmelzwasseräquivalent (in mm). Wir lesen die Datei wie üblich ein:
df = pd.read_csv("data/snow_potsdam.csv", sep=";")
df
Sieht gut aus. Lassen wir uns für den Dataframe doch mal die üblichen statistischen Kennwerte ausgeben:
df.describe()
Hm... keine Werte für die Spalte weq? Dafür aber für das Datum? Was ist da schiefgegangen??
Lösung¶
In der Spalte weq wurde ein unüblicher Wert für fehlende Daten verwendet: "novalue". Weil pandas solch einen exotischen Platzhalter nicht kennt (im Gegensatz zu z.B. nan), geht es davon aus, dass es sich bei der Spalte weq durchweg um Strings handelt. Bei der Datumsspalte hingegen müssen wir erstmal ansagen, wie dieses zu interpretieren ist.
df = pd.read_csv("data/snow_potsdam.csv", sep=";", na_values="novalue")
df.datum = pd.to_datetime(df.datum, format="%Y%m%d")
df.describe()
df
Aufgabe: Ärger in der Besenscheune¶
"Brooms Barn" ist eine landwirtschaftliche Forschungsstation in England. Die Datei ../data/brooms_barn.txt enthält Bodenkennwerte für eine dortige Versuchsfläche. Versuche, die Datei als DataFrame einzulesen. Welche Probleme treten auf? Kannst Du sie beheben?
Lösung¶
Es liegt ein so genanntes "fixed width format" vor, es gibt also keinen Spaltentrenner im bekannten Sinne. Freundlicherweise kann pandas damit umgehen, aber nur mit der Funktion pandas.read_fwd. Obendrein muss man noch bekanntgeben, dass die ersten 20 Zeilen ignoriert werden müssen und es keinen Header gibt.
df = pd.read_fwf("../data/brooms_barn.txt", skiprows=20, header=None)
df.columns = "x", "y", "K", "log10K", "pH","P", "log10P"
df
Optional: Spezialformate¶
Eigentlich könnte alles so einfach sein...es gibt standardisierte Dateiformate für so ziemlich jeden denkbaren Anwendungsfall. Und um diese Formate zu lesen, gibt es Pakete und Funktionen. So wie das altbekannte csv-Format (hier ein Beispiel von $C_{org}$-Werten an vier Punkten im Raum:
x;y;Corg
12.5;20.1;1.5
14.7;9.3;2.2
13.2;12.2;2.1
14.1;15.3;1.0Aus unerfindlichen Gründen entscheiden sich aber manchmal Menschen, ihre Daten einfach "anders" abzuspeichern. Wenn sie so freundlich sind, die Nutzung der Daten zu erlauben, nimmt man das meist stillschweigend hin.
Die Daten von oben habe ich einfach mal wie folgt in der Datei ../data/miniexample.txt abgespeichert:
x
12.5 14.7 13.2 14.1
y
20.1 9.3 12.2 15.3
Corg
1.5 2.2 2.1 1.0pandas kann das so nicht verstehen, aber wir wollen trotzdem einen Dataframe daraus machen. Was tun? Wir müssen uns eine eigene Funktion schreiben.
# Old school
def read_weirdo_format(weirdofile):
# Datei öffnen
f = open(weirdofile)
# Datei lesen
x = f.read()
# Datei schließen
f.close()
# Ggf. letzten Zeilenumbruch entfernen
x = x.strip("\n")
# Text entlang der verbleibenden Zeilenumbrüche zerschneiden
x = x.split("\n")
# Jede zweite Zeile ist ein Spaltenname
columns = x[::2]
# Jede andere zweite Zeile sind Daten
rows = x[1::2]
# Hierin wollen wir die Daten ablegen
data = []
for row in rows:
# Jede Zeile wird entlang der Leerzeichen zerschnitten
data.append(row.split(" "))
# Aus der Liste von Strings machen wir einen Array
array = np.array(data).astype("float")
# Aus dem transponierten Array machen wir einen Dataframe
df = pd.DataFrame(array.T, columns=columns)
return(df)
read_weirdo_format("../data/miniexample.txt")
Naja, geht bestimmt auch eleganter...aber es funktioniert. Alternativ können wir auch read_csv zweckentfremden.
# Dreister Missbrauch von Funktionen
def read_weirdo_format(weirdo_file):
df = pd.read_csv(weirdo_file, sep=" ", skiprows=[0,2,4], header=None).transpose()
df.columns = pd.read_csv(weirdo_file, skiprows=[1,3,5], header=None)[0]
return(df)
read_weirdo_format("../data/miniexample.txt")
Noch eine Kleinigkeit: xml, json, kml, geojson, shp, tiff, png, jpg, yaml, netcdf, hdf5, usw¶
Mit dieser Lektion haben wir gerade mal an der Oberfläche gekratzt. Es gibt so viele Datenformate, dass man leicht den Überblick verlieren kann...
- Bilddaten (Bitmaps/Raster), z.B.
- jpg
- bmp
- png
- tiff
- ...
- Geodaten (Raster):
- geotiff
- ESRI ASCII raster
- bip, bil
- ...
- Geodaten (Vektor):
- shp
- geojson
- kml
- ...
- Hierarchische und strukturierte Textformate, z.B.
- json
- xml
- yaml
- ...
- Hierarchische Formate für multi-dimensionale Arrays
- NetCDF
- HDF5
Das NetCDF-Format nehmen wir uns in Lektion 5 vor, die Geodatenformate in Lektion 6.
Ansonsten gilt stets: Falls Ihr es mit einem Euch unbekannten Dateiformat zu tun habt, dann sucht nach einer Lösung.
Optionale Aufgabe: json Lesen¶
Die Datei ../data/biowetter.json kommt vom Open Data Server des DWD und fasst die Gefahreneinschätzung des DWD für Wetterfühlige zu einem bestimmten Zeitpunkt zusammen.
Findet heraus, wie Ihr die Datei ../data/biowetter.json in Python einlesen könnt.
Lösung¶
import json
f = open("../data/biowetter.json", "r")
result = json.load(f)
f.close()
type(result)
Die Variable result ist vom Typ dict, also ein Dictionary. Ein dict kann nach Schlüsselwörtern (keys) abgefragt werden.
print(result.keys())
result["author"]
Die eigentlichen Daten liegen unter den Schlüssel zone, und zwar als Liste (list).
type(result["zone"])
len(result["zone"])
Die elf Elemente der Liste bestehen wiederum aus dicts. Wir bauen eine kleine Schleife, um uns einen Überblick über die Zonen zu verschaffen:
for i, item in enumerate(result["zone"]):
print(i, ":", item["id"], "-", item["name"])
Aha, wir befinden uns also in Zone E, die wir mit dem Index 4 aus der Liste extrahieren können. Nun schauen wir mal, ob eine Erklärung für eine heutige, wetter-bedingte unzureichende Leistungsfähigkeit im Kurs vorliegt.
print(result["zone"][4]["name"])
print("Heute nachmittag ('today_afternoon')")
print(" ", result["zone"][4]["today_afternoon"]["effect"][0]["subeffect"][0]["name"], end=": ")
print(result["zone"][4]["today_afternoon"]["effect"][0]["subeffect"][0]["value"])
print(" Empfehlungen?")
for item in result["zone"][4]["today_afternoon"]["recomms"]:
print(" ", item["name"], ":", item["value"])
Na dann.
Fazit: JSON-Daten (und ähnlich XML-Daten) sind sehr strukturiert, haben aber auch einen sehr starken Overhead durch redundante Schlüsselwörter. Man muss außerdem die Struktur gut kennen(lernen), um die Daten sinnvoll verarbeiten zu können.
Coding-Werkstatt¶
Nachsitzen¶
Hast Du noch nicht alle Aufgaben aus dieser Lektion geschafft? Dann versuche doch, diese noch nachzuarbeiten. Falls es nicht klappt, nimm die ausklappbaren Lösungsvorschläge zur Hilfe.
GRDC-Daten einlesen¶
Die Datei pvm_6457010.txt beinhaltet Abflussdaten der Oder am Pegel Gozdowice. Die Datei stammt vom "Global Data Runoff Center" (GRDC). Schreibe eine möglichst allgemeingültige Funktion, mit der Du aus der Datei folgende Daten in einen DataFrame überführst.
- Stationsname
- Längengrad und Breitengrard
- Einzugsgebietsgröße
- Höhe
- Anzahl der Jahre mit verfügbaren Daten
- Für den Gesamtzeitraum: LQ, MQ_1, MQ_2, MQ_3, HQ
Dir könnten dabei folgende Funktionen für den Vergleich von Zeichenfolgen helfen: startsWith, endsWith, identical.
Teste, ob Dein Code auch mit einer anderen Datei (pvm_6457100.txt) funktioniert.